Linus Pauling
pensava che le malattie umane riguardanti l''emoglobina, come
l'anemia falciforme, possono essere ricondotte ad una variazione
della struttura proteica della molecola di emoglobina. Per verificare
la sua ipotesi si servì dell'elettroforesi, metodo per osservare il
comportamento delle molecole dell'emoglobina di individui omozigoti
non affette dalla malattia, di omozigoti recessivi con la malattia e
di eterozigoti per l'allele dell'anemia falciforme, le quali,
disciolte in una soluzione e sottoposte ad un debole campo elettrico,
si comportano in maniere totalmente diverse. I risultati possono
essere schematizzati in questo grafico:
domenica 28 aprile 2013
L'RNA ha una struttura molecolare molto simile a quella del DNA
In
che modo la disposizione delle basi azotate nel DNA può determinare
la sequenza degli amminoacidi di una proteina?
A ciò
interviene l'RNA o acido ribonucleico, il quale possiede alcune
caratteristiche che lo differenziano dal DNA:
Esso
“traduce” le sequenze dei segmenti di DNA nelle esatte sequenze
degli amminoacidi che determinano le varie strutture proteiche,
soprattutto nel citosol.
Dopo
vari esperimenti si vide che esistono tre tipi di RNA che fungono da
intermediari nella sintesi proteica dal DNA al citosol.
Mediante il processo di trascrizione viene sintetizzato l'RNA messaggero
Durante
la fase iniziale, la RNA polimerasi riconosce delle strutture
nucleotidiche del DNA (promotori) e si attacca alla catena tramite ad
una proteina (fattore sigma); dopodiché il fattore sigma si stacca e
l'RNA polimerasi scorre lungo la doppia elica del DNA, separando i
due filamenti.
Nella fase di allungamento, utilizzando
solamente uno dei due filamenti di DNA, il filamento di stampo, l'RNA
polimerasi sintetizza il trascritto di mRNA, aggiungendo nucleotidi
che sono presenti nella cellula sottoforma di nucleosidi trifosfati
(NTP), i quali si aggiungono alla catena di RNA perdendo due gruppi
fosfato; l'mRNA fabbricato è complementare al filamento di stampo,
mentre è identico al filamento di DNA non utilizzato tranne che per
la T al posto dell'U.
Il processo termina quando si incontrano delle
strutture nucleotidiche (sequenze di arresto), bloccando così la
trascrizione.
Questo
processo è utile perché il DNA, essendo la copia matrice delle
informazioni genetiche, viene utilizzato più volte, mentre l'mRNA
deve trasportare il messaggio codificato dal DNA, attraversando il
citoplasma della cellula e, quando il suo compito termina, si
scompone per dare origine ad altre molecole.
La maturazione dell'mRNA avviene mediante lo splicing
Prima che la trascrizione sia completata, l'RNA
messaggero aggiunge un nucleotide chiamato cappuccio
(7-metilguanosina) all'estremità 5', tramite un processo denominato
capping.
Dopodiché il pre-mRNA per diventare maturo deve
sottoporsi ad un processo denominato splicing, attraverso il quale
avviene il taglio degli introni e la ricongiunzione degli esoni: per
far ciò, interviene un grosso complesso molecolare chiamato
spliceosoma, formato da subunità denominate snRNP (small nuclear
Ribo-Nucleo-Protein), ovvero delle piccole riboproteine nucleari.
Negli esseri umani (non solo) il 70% dei geni è
rielaborato in modi diversi (splicing alternativo), eliminando
introni ed esoni in modo differenti, producendo diversi tipi di
proteine.

Infine, dopo lo splicing, viene aggiunta una sequenza
nucleotidica formata solamente da adenina, chiamata coda poli-A
(conferisce stabilità al mRNA maturo, garantendone maggior durata
nel citosol).
La decifrazione del codice genetico
 |
| Severo Ochoa |
Il biochimico di origine spagnola Severo Ochoa aveva
sviluppato (premio nobel per la medicina 1959) un metodo per
sintetizzare un lungo filamento di DNA mediante l'unione di
nucleotidi; con questo metodo aveva creato un filamento di RNA in
provetta costituita da una sola base azotata, l'uracile, denominando
così il filamento “poli-U”.
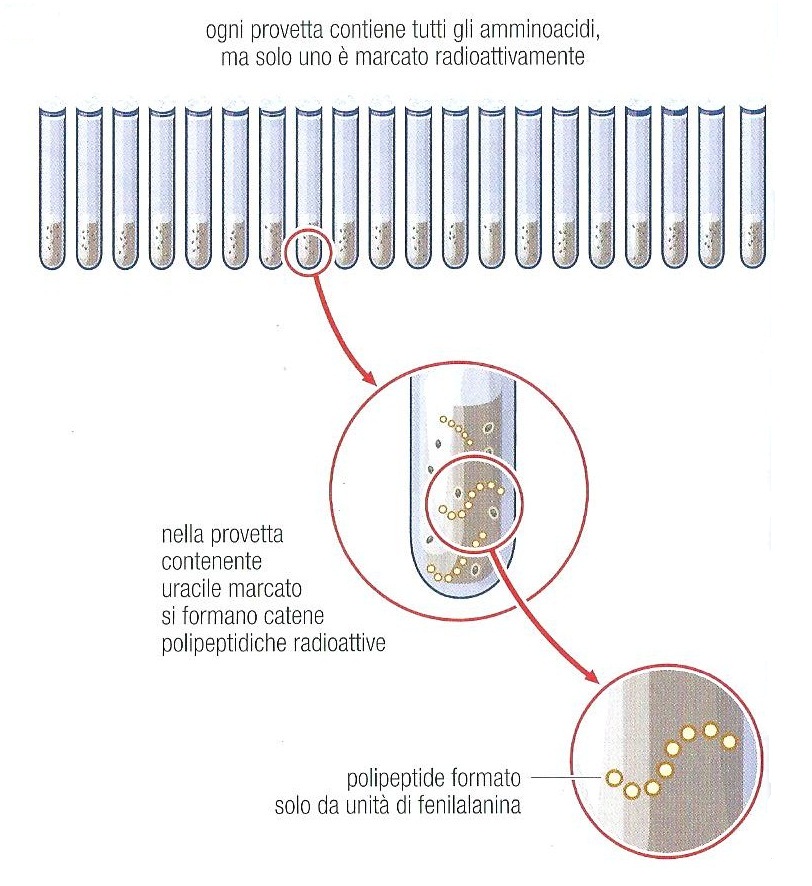
Così due scienziati, il biochimico statunitense
Marshall Warren Nirenberg e il tedesco Heinrich Matthaei, nel
1961,
prepararono 20 provette contenenti ciascuna ribosomi, ATP, enzimi e
amminoacidi, ognuno dei quali, e uno solo, era marcato
radioattivamente. In 19 provette non si produsse nessun polipeptide,
ma nella restante, in cui era presente la fenilalanina radioattiva,
si formarono catene polipeptidiche UUU.
Esperimenti simili furono fatti nel corso del tempo da
altri scienziati, cosicchè furono decifrati tutti i possibili codoni
del RNA messaggero. Delle 64 combinazioni possibili di triplette, 3
sono segnali di arresto, mentre le restanti 61 codificano gli
amminoacidi corrispondenti.
Ma come mai esistono 64 combinazioni possibili per
codificare i venti amminoacidi?
Gli scienziati ebbero identificato l'mRNA come la copia
di lavoro delle istruzioni genetiche che il DNA doveva eseguire per
fabbricare le proteine, ma si chiesero anche come il DNA potesse
codificarle. Le proteine contengono 20 amminoacidi differenti
disposti linearmente, mentre il DNA e l'RNA contengono ciascuno 4
nucleotidi disposti linearmente in sequenze differenti. Se ciascun
nucleotide codificasse un singolo amminoacido, allora le 4 basi
azotate corrisponderebbero solamente a 4 amminoacidi. Se una sequenza
di due nucleotidi codificasse invece un amminoacido, ci sarebbero 42
possibilità di risoluzione, ma gli amminoacidi sono ben 20. Perciò,
si arrivò a scoprire che ad una sequenza di tre nucleotidi,
corrispondeva un amminoacido, ottenendo 43 possibilità di
risoluzione, cioè 64. Ogni sequenza (tripletta) è definita codone.
Quando un
amminoacido è decodificato da 4 triplette che differiscono solamente
per l'ultimo nucleotide, il codice è detto degenerato.
Il
codice genetico è identico per tutti gli esseri viventi sul pianeta
terra, nel senso che se prendessimo una tripletta nucleotidica, essa
traduce un amminoacido che è identico nell'uomo, ma anche in un
fungo, oppure in un lichene...
L'unica eccezione sono i mitocondri, i quali contengono
un proprio DNA, indipendente da quello nucleare, che viene trascritto
in mRNA, sintetizzando proteine mitocondriali particolari.
Gli RNA ribosomiale e di trasporto svolgono la loro funzione nel citoplasma
La sintesi proteica è meno complessa nei procarioti, rispetto agli eucarioti, ma entrambi gli organismi
possiedono delle comunanze; una caratteristica fondamentale che
riguarda la trascrizione è che si svolge nel nucleo, mentre la sintesi proteica nel citoplasma, a differenza delle cellule procarioti.
La sintesi necessita innanzitutto di altri due tipi di
RNA: quello ribosomiale (rRNA) e quello di trasporto (tRNA).

L'RNA ribosomiale è contenuto naturalmente nei ribosomi, formati per un terzo da proteine e per due terzi da RNA; essi sono costituiti da due subunità:
- la subunità minore che possiede un sito di legame per l'RNA messaggero;
- la subunità maggiore che possiede tre siti di legame per gli RNA di trasporto.
 |
| Robert Holley |
traduce il linguaggio degli acidi nucleici in proteine, contenuto
nelle cellule in più di 20 tipi diversi, almeno uno per ciascun
amminoacido. Esso presenta una struttura a trifoglio, formata da una
catena in direzione 5' – 3'. Questo filamento è formato da basi
invariabili (ad esempio la sequenza finale CCA presso l'estremità
3') per tutti i tipi di tRNA, ma ne presenta anche di variabili:
- un anticodone formato da tre nucleotidi variabili e complementari all'mRNA;
- una regione che sintetizza un enzima chiamato amminoacil-tRNA sintetasi, il quale attira i specifici amminoacidi alle catene di RNA di trasporto, cosicché quest'ultimo può essere trasportato al sito di una formazione polipeptidica.
domenica 21 aprile 2013
Il controllo genico nei eucarioti
Il
controllo durante la trascrizione negli eucarioti presenta analogie e
differenze con il sistema dei procarioti. Infatti in entrambi gli
organismi agiscono le molecole effettrici, gli attivatori e i
repressori che influenzano l'attività di codificazione dell'RNA
polimerasi; mentre nelle DNA eucariote non esiste l'operone, perché
ogni gene strutturale possiede un proprio sistema di controllo.
La cromatina (DNA
+ proteine) è in relazione col tasso di informazione genica che si
vuole trasmettere; infatti può essere di due tipi, identificabili
attraverso la colorazione:
- l'eucromatina, ovvero la cromatina che risulta più dispersa, meno densa e che si colora debolmente. Essa si trova nell'interfase, dove permette l'accesso delle molecole di RNA polimerasi per permettere la trascrizione (perciò presenta una conformazione aperta);
- l'eterocromatina, ovvero la cromatina maggiormente condensata, compatta e che si colora più intensamente. La cromatina si trova durante questo stato durante la divisione cellulare o nei centromeri ad esempio, inibendo l'attività di trascrizione (presentando perciò una conformazione chiusa).
 |
| Corpo di Barr |
 |
| Murray Llewellyn Barr |
La trascrizione
può avvenire come si è visto solo se la cromatina è
despiralizzata, come suggeriscono i corpi di Barr, ovvero i cromosomi
X che nei mammiferi di sesso maschile sono presenti in copia unica,
ma nelle femmine in due coppie, permettendo conseguentemente la
produzione del doppio delle proteine; invece uno dei due cromosomi
sessuali è spiralizzato costantemente già dalla fase embrionale.
 |
| Istone-acetiltransferasi |
Se la cromatina
risulta inizialmente spiralizzata, come è possibile che essa si
despiralizzi momentaneamente?
Per far ciò
intervengono degli enzimi attivatori (più di 50 nei mammiferi), come
l'istone-acetiltransferasi, il quale si attacca attraverso gruppi
acetili (-COCH3) agli istoni.
Il promotore delle
cellule eucariote, a differenza di quello dei procarioti, è
costituito da tre regioni differenti:
- La prima regione è il TATA box (5' – TATAAA – 3'), sequenza che localizza il punto da cui la trascrizione potrebbe iniziare;
- Un'altra regione, il sito di inizio della trascrizione, dove l'RNA polimerasi si inserisce per avviare la trascrizione, il quale sommato al TATA box costituisce il promotore basale (core promoter), a cui sono legati 5 fattori di trascrizione generali (GTF) per consentire l'attacco della RNA polimerasi. Si viene a formare così quello che è definito complesso di pre-inizio;
- La terza regione è il sito degli elementi regolatori, formato da sequenze nucleotidiche denominate rispettivamente enhancer o silencer, a seconda della funzione (avvio o inibizione della trascrizione di un gene).
Spiegato ora com'è
costituito un promoter eucariote, bisogna dire come avviene l'inizio
del processo di trascrizione.
Un attivatore si
lega ad un enhancer, favorendo a sua volta l'attacco di un fattore di
trascrizione al TATA box e la conseguente formazione del complesso di
pre-inizio.
Capita però
alcune volte che l'enhancer si trovi lontano dalla sequenza
promotrice, perciò interviene una proteina mediatrice (mediatore)
che mette in comunicazione l'attivatore dell'enhancer con il
complesso di pre-inizio.

Il controllo genico nei procarioti
Attualmente
la trascrizione nei procarioti si basa su un modello chiamato
operone, proposto dai biologi francesi François Jacob e Jacques
Monod.
 |
| Françis Jacob |
 |
| Jacques Lucien Monod |
Secondo
tale modello, il DNA è formato in successione da una sequenza
promotrice dove si attacca la RNA polimerasi (promotore), da una
sequenza che fa da aggancio per gli enzimi attivatori o i repressori
(operatore), ed infine dai geni strutturali che codificano i futuri
polipeptidi.
Gli
attivatori o repressori (fattori di regolazione della trascrizione)
possiedono dei siti di legame per delle molecole effettrici, le quali
attaccandosi o staccandosi dal fattore danno il segnale di comando a
questi enzimi, cioè se effettuare un controllo negativo
(repressione) o uno positivo (induzione); le sequenze che codificano
le proteine di regolazione si chiamano geni regolatori, le quali si
possono trovare vicino all'operone o sparse per il cromosoma.
La
capacità di un repressore di legarsi all'operone è data perciò
dalla molecola effettrice, la quale è detta corepressore se lo
attiva, altrimenti se lo disattiva si chiama induttore. Vediamo due
esempi concreti sulla repressione:
Quando
il livello di triptofano è alto, esso funge da molecola effettrice o
meglio da corepressore, legandosi al repressore trp, inducendone la
sua attivazione, il quale poi si lega all'operatore che blocca la
trascrizione.
Invece,
quando è presente il lattosio, si instaura un meccanismo di
produzione dell'allolattosio, il quale funziona da molecola
effettrice o meglio da induttore, cioè si lega al repressore,
disattivandolo e facendolo staccare dall'operone del lattosio (lac).
Di conseguenza la RNA polimerasi trascrive tre enzimi di seguito che
serviranno per smontare il lattosio nei suoi due costituenti
(l'enzima beta-galattosidasi, la beta-galattoside permeasi, e
l'enzima transacetilasi).
Esistono
comunque proteine indispensabili per la cellula, tanto che i geni che
le codificano sono controllati con minore rigidità, essendo sempre
attivi: essi sono chiamati geni costitutivi (housekeeping).
La regolazione genica
I
geni, ovvero la porzione di DNA che codifica un polipeptide, hanno la
capacità di attivarsi e disattivarsi all'interno della cellula,
attraverso processi che fanno parte di quale controllo del genoma che
è definito regolazione genica.
 Il
vantaggio è un minor dispendio di energia da parte della cellula di
cui i geni ne fanno parte; ad esempio all'interno di un batterio di
Escherichia coli, l'enzima beta-galattosidasi scinde la molecola del
lattosio, disaccaride formato da glucosio e galattosio, solo se è
presente lo zucchero, altrimenti la loro creazione risulta inutile.
Il
vantaggio è un minor dispendio di energia da parte della cellula di
cui i geni ne fanno parte; ad esempio all'interno di un batterio di
Escherichia coli, l'enzima beta-galattosidasi scinde la molecola del
lattosio, disaccaride formato da glucosio e galattosio, solo se è
presente lo zucchero, altrimenti la loro creazione risulta inutile. |
| L'enzima beta-galattosidasi |
In altri casi, se la sostanza che gli enzimi fabbricano (esempio triptofano) è presente all'interno della cellula, allora questi si decompongono, altrimenti se il triptofano è assente, le proteine che eseguono la biosintesi per creare questo tipo di amminoacido sono indispensabili.
 |
| Triptofano |
Nei
procarioti in genere il controllo genico avviene per attivazione o
disattivazione delle unità ereditarie, mentre negli eucarioti la
faccenda è più complessa.
Lo
zigote si divide ripetutamente per mitosi e citodieresi, originando
molte cellule, le quali avranno lo stesso patrimonio genetico
(genoma), ma differente corredo proteica (proteoma), avendo ognuna
funzioni e strutture diverse.
Tutto
questo è ben visibile nei globuli rossi ad esempio: infatti, allo
stato embrionale, i globuli rossi sintetizzano un tipo di emoglobina
maggiormente affine all'ossigeno, cosa che diminuisce allo stadio di
feto tramite un secondo tipo di emoglobina e in breve tempo si
producono catene polipeptidiche come nella fase adulta.
Perciò ogni cellula del nostro corpo contiene le sequenze di DNA per codificare tutti gli enzimi, ma sono attivate e disattivate in base al tipo di cellula.
Perciò ogni cellula del nostro corpo contiene le sequenze di DNA per codificare tutti gli enzimi, ma sono attivate e disattivate in base al tipo di cellula.
Per
dimostrare ciò, il biologo J. B. Gurdon condusse un esperimento, nel
quale irradiò un ovulo
 |
| John Bertrand Gurdon |
Le mutazione geniche
Una
mutazione è un cambiamento dei nucleotidi di una sequenza
all'interno di un acido nucleico.
Quando
sono presenti nei gameti o nelle cellule che li originano, esse si
protrarranno anche alle generazioni successive, mentre le mutazioni
che agiscono nelle cellule somatiche sono trasmesse alle cellule
figlie per mitosi e citodieresi.
Le
mutazioni in genere riguardano la sostituzione, aggiunta o perdita di
un singolo nucleotide o di poche unità, e sono definite mutazioni
puntiformi, le quali si presentano in modalità diverse:
- Quando viene sostituita una base azotata, cambia l'amminoacido che è presente solitamente in una proteina, causando al polipeptide una mutazione di senso (missense mutation), come nell'anemia falciforme. La molecola difettosa di emoglobina che la caratterizza è formata da quattro catene polipeptidiche, due filamenti alfa e due beta. Le catene beta in una molecola di emoglobina normale contengono in un punto preciso una sequenza di acido glutammico (glu); mentre in un filamento beta delle cellule falciformi è presente la valina (val). Questa mutazione è avvenuta perché l'mRNA possiede la sequenza GUA (val) invece di GAA (glu), mentre nel filamento stampo del DNA da cui viene trascritto l'mRNA risulta essere GUG (val) invece delle sequenza GAG (glu).
- Un altro tipo di mutazione che avviene per sostituzione di una base azotata è definita mutazione non senso (nonsense mutation), la quale consiste nella sostituzione di una sequenza nucleotidica normale con una di arresto, causando lo “stop” della sintesi proteica prima del tempo. Le conseguenze sono maggiormente gravi, come la distrofia muscolare di Duchenne, venendo a mancare la proteina distrofina (fondamentale per il tessuto muscolare).
- Un terzo tipo di mutazione puntiforme è detta silente (silent mutation), nella quale la sostituzione di un nucleotide non comporta il cambiamento della proteina: infatti la tripletta AAA dell'mRNA cosi come la tripletta AAG codifica sempre l'amminoacido lisina (lys).Oltre alla sostituzione può avvenire la perdita (delezione) o aggiunta (inserimento) di un nucleotide, cambiando il modo di lettura dei nucleotidi, producendo spesso proteine nuove e non funzionanti.
Le
mutazioni, viste macroscopicamente, possono essere spontanee o
indotte.
lunedì 8 aprile 2013
Anche il processo di traduzione avviene in tre fasi successive
Il processo di
traduzione o di sintesi proteica ha il compito di elaborare e
decodificare il messaggio nucleotidico dell'mRNA nel linguaggio degli
amminoacidi delle proteine.
Esso consiste in
tre fasi principali:
- la fase d'inizio comincia con l'attaccamento della subunità minore del ribosoma al filamento dell'RNA messaggero. In seguito il tRNA si attacca per mezzo dell'anticodone (solitamente 3'-UAC-5') complementare al primo codone dell'mRNA (conseguentemente 5'-AUG-3'), portando con sé l'amminoacido modificato della metionina, la formilmetionina (fMet). Dopodiché la subunità maggiore del ribosoma, formata da tre siti specifici, si attacca a quella minore, causando l'occupazione del sito P (polipeptide) del tRNA.
- la fase di allungamento comporta l'attaccamento di un altro tRNA con anticodone complementare al secondo codone del RNA messaggero, occupando il sito A (amminoacile)della parte superiore del ribosoma. Si forma un legame peptidico fra i due amminoacidi, ricavando energia dal nucleotide GTP (guanosina trifosfato) del secondo RNA di trasporto. Poi l''mRNA, scorrendo in avanti, provoca lo spostamento del primo tRNA nel sito E (espulsione), il quale viene liberato, mentre il tRNA seguente slitta in P, dando lo spazio ad un terzo tRNA di insediarsi nel sito A, ripetendo l'operazione altre svariate volte.
- la fase di terminazione determina l'arresto della sintesi proteica, a causa di un codone di arresto, il quale, non possedendo nessun tRNA complementare, accoglie una proteina detta fattore di rilascio, dissociando le subunità del ribosoma e rimuovendo la catena polipeptidica.
Ricorda!
I ribosomi possono
sintetizzare e fabbricare polipeptidi contemporaneamente con stesso
mRNA, e vengono chiamati polisomi o poliribosomi.
Le proteine
prodotte, in base alle sequenza di amminoacidi che le caratterizzano
(sequenza segnale), si dirigono nel citosol, e gran parte di esse
entrano nel nucleo o nei mitocondri; altri proteine (enzimi digestivi
e gli ormoni) vengono indirizzate attraverso l'apparato di Golgi dal
reticolo endoplasmatico, nel quale le biomolecole vengono rielaborate
e “impacchettate” in vescicole espulse fuori dalla cellula o in
un compartimento cellulare.
Iscriviti a:
Post (Atom)












.jpg)








